Fakultät für Informatik - Technische Universität München
Lehrstuhl für Effiziente Algorithmen

Fakultät für Informatik - Technische Universität MünchenLehrstuhl für Effiziente Algorithmen |
 |
 |
Hauptseminar Bioinformatik im SS 2004:
von Genexpressionsdaten aus Microarrays |
 |
|||
|
Termin: Dienstags, 14:15 - 15:45
Ort: Informatik-Gebäude, Raum 03.07.023 |
||||||||
| Kontakt: Jens Ernst , Raum 03.13.061, Telefon: 289-19426 | ||||||||
|
|
||||||||
| Überblick:
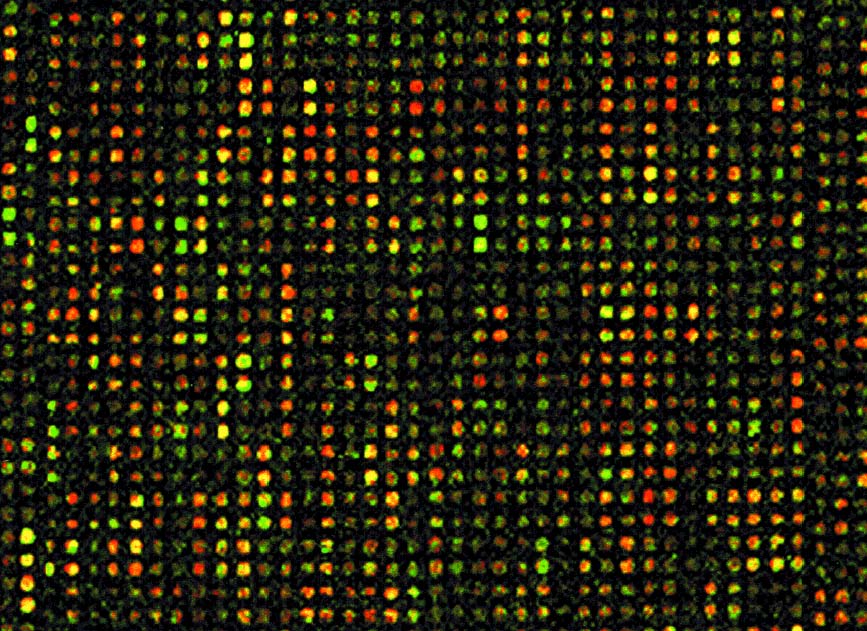
DNA-Microarrays sind eine relativ neue, sehr effiziente Technologie zur experimentellen Analyse des Expressionsverhaltens von Genen. Mit ihrer Hilfe läßt sich die Aktivität tausender Gene parallel beobachten. Hierbei wird ausgenutzt, daß einsträngige Nucleinsäuren Hybridisierungsreaktionen eingehen, d.h. in einer heterogenen Mischung verschiedener DNA oder RNA-Moleküle einen komplementären Partner finden und chemisch an diesen binden können. DNA-Chips werden oft mit Hilfe von Spotting-Robotern (siehe oben) hergestellt, die zu einzelnen Genen gehörige DNA-Stücke auf einer geeignet präparierten Glasscheibe unterbringen. Mehrere tausend Gene können auf ein Array gespottet werden. Die DNA-Stücke sind hierbei mit fluoreszenten Markierungen versehen. Das gesamte Array wird einer Hybridisierung unterworfen, zum Beispiel mit einer Gewebeprobe, wodurch ermittelt werden kann, in welcher Konzentration die mRNA der auf dem Chipbefindlichen Gene in der Probe enthalten sind. Dies wird verwendet als Maß für die Expression dieser Gene unter den äußeren Bedingungen, unter denen die Probe entstanden ist. Die Messung der Expressionsintensität erfolgt durch Bestrahlung des Chips mit Laserlicht und Scannen. Üblicherweise wird rotes und grünes Licht verwendet, wodurch man eines der typischen rot-grün-gelb-Microarraybilder erhält (siehe oben). Führt man für mehrere Microarrays mit gleicher Bestückung nun Hybridisierungsexperimente durch, so erhält man für jedes der Gene eine Folge von charakteristischen Expressionswerten, die auch als Fingerabdruck (fingerprint) bzw. Expressionsprofil des jeweiligen Gens bezeichnet werden. Ziel des Vergleichs der Expressionsprofile vieler (evtl. sogar aller) Gene eines Organismus sind häufig Rückschlüsse auf die Funktion dieser Gene. Dieses Hauptseminar beschäftigt sich mit den Grundlagen der Genexpressionsanalyse durch DNA-Microarrays aus Datensicht. Nach einem Überblick über die biolo- gischen Hintergründe sowie die Mircoroarraytechnologie werden Fragen der Normalisierung von Microarray-Daten und algorithmische Probleme der Strukturfindung betrachtet. Von besonderem Interesse ist hier das Clustering-Problem. Bei allen vorgestellten Verfahren steht die Problematik schlechter Datenqualität im Hintergrund. |
||||||||
|
|
||||||||
| Vorträge: | ||||
Weitere Termine werden ggf. noch bekanntgegeben |
||||
|
|
||||